日本政府は、先日オミクロン株が検出された場合、“濃厚”接触者(単に同じフライトに搭乗しているだけで濃厚??)とみなして、指定の施設に隔離すると発表しています。

昨日オミクロン株(スパイクタンパク質領域の遺伝子の複数の変異)をどうやって検出しているか不思議だという内容をお伝えしました。
遺伝子解析をかけるためには、スパイクタンパク質の遺伝子領域をPCRと同じ原理で、増幅する作業が最初に必要となります。

このときに使用するプライマーという遺伝子配列(oligomer)に当初からは改良が加えられているものの、それでもオミクロンのような変異型を検出できない場合があることが報告されています(『Large swathes of dark-matter (not-sequenced) in the SARS-Cov2 spike protein in significant number of samples in GISAID – probably due to ARTIC-primer artifacts – which will mask real mutations in these genomic regions, and where/when some mutations arose.』 10.31219/osf.io/hukgm)(Analysis of the ARTIC version 3 and version 4 SARS-CoV-2 primers and their impact on the detection of the G142D amino acid substitution in the spike protein. bioRxiv preprint doi: https://doi.org/10.1101/2021.09.27.461949)。
遺伝子解析でも、捉えられない遺伝子領域(スパイクタンパク質を産生する遺伝子配列)があることも示されています。
遺伝子解析でも正確に検出できないのは、特にウイルスの量が少ない場合(low amplicon)に顕著になります(Improvements to the ARTIC multiplex PCR method for SARS-CoV-2 genome sequencing using nanopore. Version 1. bioRxiv. Preprint. 2020 Sep 4.doi: 10.1101/2020.09.04.283077)(A proposal of alternative primers for the ARTIC Network’s multiplex PCR to improve coverage of SARS-CoV-2 genome sequencing. BioRxiv [Internet]. biorxiv.org; 2020; Available from: https://www.biorxiv.org/content/10.1101/2020.03.10.985150v2.abstract)。

つまり、遺伝子解析もまだまだ改良の余地のある、発展途上の検査法であるということです。
したがって、オミクロン疑いで遺伝子解析をかけても、完全に遺伝子配列が明らかになるということではないことだけは確認できました。
2020年9月の時点で、すでにデータベース(GISAID)にあがっている新型コロナウイルスの遺伝子配列は9万種類を超えています。
現在ではさらに多くの種類の新型コロナウイルスが報告されているはずです。
現在、オミクロンと騒いでいるのは、今まで遺伝子解析で検出できなかった変異型が検出できるようになっただけではないかと推測しています。

なぜなら、南アフリカから短期間で急激に世界に拡大するということはあり得ないからです。

ちなみに、オミクロン変異が最初に発見された南アフリカの症例は、データベースからは2021年6月17日となっています(現在、この情報はデータベースから削除されている)。
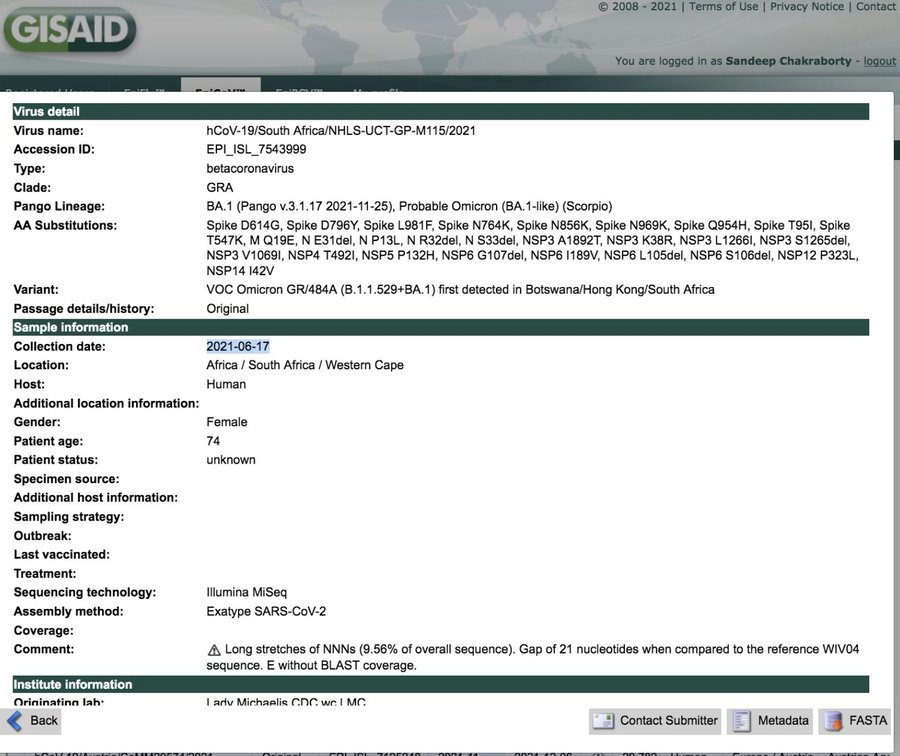
感染力の高いとされているオミクロンが5ヶ月待って世界に拡大するでしょうか?
オミクロンとされている変異型のオリジナルも5ヶ月も経過すれば、すでに数百種類のオミクロン変異型となっているでしょう。
新型コロナパンデミックがPCRパンデミックであると当初からお伝えしていたように、オミクロンなどの変異型騒ぎも遺伝子解析パンデミックという検査の不正確さがもたらした(不正確さを利用した)お祭り騒ぎであることは、今後明らかになってくるでしょう(^_−)−☆。